구글의 이미지 인식 딥러닝 인공지능 AI로 암과 종양의 식별이 가능하다
'Nature Medicine'에 발표된 연구에 따르면 뉴욕대학교(NYU)의 연구원들은 구글의 기존 딥러닝 알고리즘을 재교육하여 주요 2종류의 폐암을 97%의 정밀도로 판별할 수 있게 되었다. 이런 종류의 인공지능은 이미지로 얼굴, 동물, 물체 등을 식별하는 구글의 온라인 서비스에 사용되는 것과 동일한 기술을 사용하고 있다.
인간 병리학자들은 사진으로 종양을 식별하기 위해 엄청난 훈련을 받는다. 이번의 NYU의 신경회로망은 지금까지 병리학자가 할 수 없었던 방법도 학습했다고 한다. 1장의 사진에서 각 종양에 유전자 변이가 대량 발생하고 있는지 여부를 확인하였다. 이에 대한 논문저자 Aristotelis Tsirigos는 이번 인공지능이 인간 의사만큼 뛰어나다는 것보다 새로운 통찰력을 제공할 수 있다는 점이 중요하다고 말했다.
이번 연구를 위해 Tsirigos의 연구팀은 구글의 알고리즘이 먼저 1000 종류의 물체를 식별하도록 훈련했다. 'Inception v3'라는 오픈소스 알고리즘을 사용하였다. 암 조직과 건강한 조직의 이미지는 'The Cancer Genome Atlas (TCGA)'가 공개하는 환자의 조직 샘플 라이브러리에서 확보하였다. 이를 통해 얻은 수십만장의 암 조직의 이미지를 알고리즘에게 보여주었다.
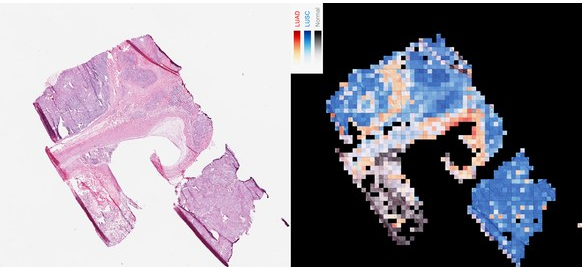
Inception가 암 세포를 99%의 정확도로 식별할 수 있게 되자, 다음 단계로 선암종(Adenocarcinoma, LUAD)과 편평세포암종(squamous cell carcinoma, LUSC)의 2종류의 폐암을 구별하도록 학습시켰다. 두 종류는 모두 가장 많이 발병하는 폐암이다. 현미경으로 보면 비슷하지만 치료법이 다르다.
시리고스 연구팀은 다시 TCGA 데이터를 이용하여 Inception에 슬라이드 사진과 함께 각각의 종양의 유전자 프로파일을 주었다. 새로운 이미지로 실험을 실시했는데, 어떤 이미지에 암 조직이 나타나고 있는지를 식별할 뿐만 아니라 특정 조직 샘플의 유전자 변이도 식별 할 수 있었다.
연구팀은 인공지능을 훈련시키기 위해 인간 병리학자의 도움을 구했었다. 처음에는 프로젝트 참여를 두려워했으나 협력자를 찾는 것은 쉬웠다. 의사들은 Inception이 무엇을 할 수 있는지 궁금해 하였다. 병리학자들은 폐암뿐만 아니라 자신들의 프로젝트를 해결할 방법도 궁금해했다.
저자 시리고스는 인간 의사를 기계로 완전히 대체할 수는 없으리라고 말했다. 기계는 간단한 문제에 대응하고 병리학자들은 더 깊은 문제에 대응할 수 있다. 물체 인식을 기계에 맡겨도 인간만이 할 수 있는 의료범위는 충분히 남아있다.
출처 : Wired, GOOGLE AI TOOL IDENTIFIES A TUMOR'S MUTATIONS FROM AN IMAGE
발췌 : In new research published today in Nature Medicine, scientists at New York University re-trained an off-the-shelf Google deep learning algorithm to distinguish between two of the most common types of lung cancers with 97 percent accuracy. This type of AI—the same tech that identifies faces, animals, and objects in pictures uploaded to Google’s online services—has proven adept at diagnosing disease before, including diabetic blindness and heart conditions.
논문 : Nature Medicine, Classification and mutation prediction from non–small cell lung cancer histopathology images using deep learning
Abstract : Visual inspection of histopathology slides is one of the main methods used by pathologists to assess the stage, type and subtype of lung tumors. Adenocarcinoma (LUAD) and squamous cell carcinoma (LUSC) are the most prevalent subtypes of lung cancer, and their distinction requires visual inspection by an experienced pathologist. In this study, we trained a deep convolutional neural network (inception v3) on whole-slide images obtained from The Cancer Genome Atlas to accurately and automatically classify them into LUAD, LUSC or normal lung tissue. The performance of our method is comparable to that of pathologists, with an average area under the curve (AUC) of 0.97. Our model was validated on independent datasets of frozen tissues, formalin-fixed paraffin-embedded tissues and biopsies. Furthermore, we trained the network to predict the ten most commonly mutated genes in LUAD. We found that six of them—STK11, EGFR, FAT1, SETBP1, KRAS and TP53—can be predicted from pathology images, with AUCs from 0.733 to 0.856 as measured on a held-out population. These findings suggest that deep-learning models can assist pathologists in the detection of cancer subtype or gene mutations. Our approach can be applied to any cancer type, and the code is available at https://github.com/ncoudray/DeepPATH
댓글 없음:
댓글 쓰기